С развитием пандемии COVID-19 и эволюцией вируса SARS-CoV-2 уже в конце 2020 г. стали появляться сообщения о появлении его новых генетических вариантов [1]. Первоначально эти варианты обозначались как британский и африканский по названию страны происхождения. Впоследствии они были обозначены Альфа и Бета после введения ВОЗ классификации геновариантов коронавируса SARS-CoV-2 с обозначением буквами греческого алфавита [2]. Оба этих варианта достаточно быстро распространились в Великобритании и странах Африки, а затем начали активно распространяться и на территории других стран [3–7].
Отличием вариантов Альфа и Бета от доминировавшего до их появления варианта вируса SARS-CoV-2 (содержащего мутацию D614G в S-белке) является присутствие дополнительных мутаций в различных областях генома коронавируса, 8 таких мутаций расположены в области S-белка (табл. 1). Характерными мутациями геноварианта Альфа являются замены N501Y, A570D и делеции del_HV69-70 и del_Y144, а для варианта Бета – замены D80A, E484K, N501Y и делеция del_LLA241-243. В литературе высказывается предположение, что роль замены N501Y состоит в усилении связывания S-белка коронавируса SARS-CoV-2 с ACE2-рецептором человека, что облегчает проникновение вируса в клетку и, как следствие, способствует повышению его трансмиссивности и более активному распространению в человеческой популяции [8–10].
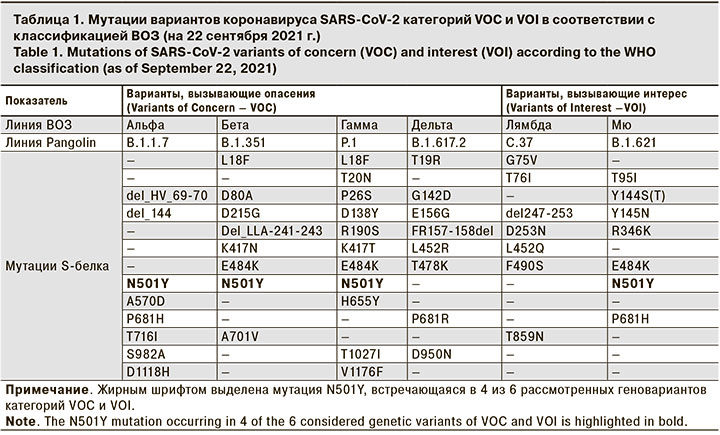
ВОЗ постоянно ведет наблюдение за распространением генетических вариантов SARS-CoV-2 и регулярно формирует список таких вариантов с делением на 3 группы: варианты, вызывающие опасения (Variants of Concern – VOC); варианты, вызывающие интерес (Variants of Interest – VOI) и варианты, требующие наблюдения (Variants Under Monitoring – VUM) [11].
К концу сентября 2021 г. в список VOC-вариантов входили Альфа, Бета, Гамма, Дельта; в список VOI-вариантов – Лямбда, Мю, а в список VUM-вариантов – еще 14 геновариантов. Списки мутаций в S-белке для вариантов VOC и VOI приведены в табл. 1, из которой видно, что 4 из 6 актуальных геновариантов содержат мутацию N501Y.
Несмотря на доминирование линии Дельта на территории Российской Федерации с июня 2021 г. по настоящее время (конец октября 2021 г.) [12], разработка простых и недорогих скрининговых методов детекции точечных мутаций вируса SARS-CoV-2 является актуальной задачей. Такая система скрининга позволит снизить трудозатраты и финансовые расходы на секвенирование в рамках изучения генетического разнообразия вируса и позволит контролировать появление или завоз новых геновариантов. На примере выявления мутации N501Y мы сравним использование 3 методов: секвенирования методом Сэнгера, ПЦР в реальном времени (ПЦР-РВ) и петлевой изотермической амплификации (Loop Mediated Isothermal Amplification – LAMP).
Мониторинг за генетическим разнообразием вируса SARS-CoV-2 проводится во всех странах. Основным методом, который используется для решения этой задачи, в настоящий момент является секвенирование. Полногеномное секвенирование позволяет получить максимум информации об образце и максимально достоверно определить принадлежность к той или иной генетической линии. Однако существенным его недостатком является высокая стоимость и достаточно длительный (до 4 дней) и сложный процесс подготовки проб.
Секвенирование отдельных фрагментов генома вируса с целью генотипирования проводят, как правило, методом Сэнгера [13]. Это позволяет снизить стоимость процесса, однако так же, как и полногеномное секвенирование, требует достаточно длительной процедуры пробоподоготовки и проведения секвенирования (3 дня от поступления пробы до получения результата). Кроме стадии выделения РНК вируса и проведения обратной транскрипции для получения кДНК при секвенировании необходимо проводить амплификацию целевых фрагментов генома, их очистку и затем уже пробоподготовку собственно для секвенирования по стандартным протоколам. Неоспоримым достоинством данного метода является высокая точность определения генетической последовательности и более низкая стоимость по сравнению с методами полногеномного секвенирования.
Следует отметить, что и полногеномное секвенирование, и фрагментное секвенирование методом Сэнгера чувствительны к качеству биоматериала, особенно если размеры получаемых ампликонов составляют больше 500 пн. При транспортировке, хранении и повторяющихся циклах заморозки–разморозки образцы, особенно в случае РНК-содержащих вирусов, могут значительно деградировать, что затруднит или сделает невозможным получение результатов методами секвенирования.
Отдельную нишу среди методов детекции точечных мутаций занимают методы на основе ПЦР [14–16]. Для этой цели широко используется такой хорошо известный вариант метода, как ПЦР-РВ. Такие разработки описаны, в том числе и для детекции мутаций коронавируса [17, 18]. Неоспоримым преимуществом ПЦР-РВ является высокая чувствительность и специфичность. Также стоит отметить существенно более высокую скорость получения результатов по сравнению с методами секвенирования. Также экономия времени при проведении ПЦР-РВ возможна за счет реализации технологии одноступенчатой амплификации при объединении стадий обратной транскрипции и амплификации ДНК в одной пробирке (ОТ-ПЦР) [19].
Еще большее снижение временных затрат возможно при использовании изотермической амплификации (ИТ). В последнее время данный метод активно применяется для детекции патогенов [20], в том числе вируса SARS-CoV-2 [21–25]. Основным преимуществом метода является скорость. Длительность реакции снижается за счет проведения ИТ при одной температуре, не тратится время на нагрев и охлаждение смеси. Другими существенными отличиями метода являются использование нескольких пар диагностических праймеров и применение ДНК-полимеразы с вытесняющей активностью. В среднем экономия времени при замене ПЦР-РВ на ИТ может составлять до 20–30 мин. Также возможно проведение ИТ в одну стадию с обратной транскрипцией при детекции РНК-содержащих мишеней (ОТ-ИТ). В последнее время появляются публикации, в которых описано использование ИТ для детекции точечных мутаций.
Одним из самых распространенных способов обнаружения SNP (Single Point Mutation) с помощью ИТ является технология, разработанная Eiken Chemical Co., Ltd. [26]. Она заключается в использовании внутренних праймеров FIP и BIP, несущих SNP на своих 5’-концах (рисунок, см. на вклейке). Такой подход с различными модификациями описан в многочисленных публикациях [27, 28] и в своем первоначальном варианте не отличается по количеству используемых праймеров от стандартной ИТ.
Следует отметить, что возможность использования разных методов детекции в сочетании с возможностью упрощенной пробоподготовки и достаточно быстрым получением результатов (от 30 мин до 1 ч) делают ИТ перспективным скрининговым методом для применения в полевых условиях или в формате тестирования у постели больного.
Материалы и методы
В настоящем исследовании в качестве источника генетического материала коронавируса SARS-CoV-2 использовались мазки из рото- и носоглотки от пациентов с симптомами новой коронавирусной инфекции и подтвержденным методом ПЦР-РВ диагнозом COVID-19 (372 образца). Материал помещали в транспортную среду для мазков («АмплиСенс», Россия). Выделение РНК из 100 мкл клинического материала проводили с помощью набора реагентов «РИБО-преп» («АмплиСенс», Россия). Далее 20 мкл выделенной РНК использовали для получения тотальной кДНК, применяя набор реагентов «РЕВЕРТА-L» («АмплиСенс», Россия). Наличие генетического материала SARS- CoV-2 в мазках было подтверждено методом ПЦР-РВ с использованием набора реагентов «АмплиСенс COV-Bat-FL» («АмплиСенс», Россия). Результаты детектировали в соответствии с инструкциями производителя на приборе ДТ-96. Полученные данные анализировали в соответствии с инструкциями производителя в программе RealTime PCR v.7.9, поставляющейся с прибором («ДНК-Технология», Россия).
Для исследования отбирали клинические образцы, в которых пороговый цикл (Ct) при ПЦР-анализе не превышал 20. Низкий показатель Ct соответствует высокой вирусной нагрузке и обеспечивает достаточное количество генетического материала вируса в пробе, что является важным фактором для анализа проб методом секвенирования по Сэнгеру.
Обнаружение мутации N501Y в S-белке коронавируса проводили 3 методами: секвенированием по методу Сэнгера, ПЦР-РВ с применением собственной разработанной in-house методики и методом ИТ с использованием набора реагентов «АмплиСенс®SARS-CoV-2-N501Y-IT» (РУ № РЗН 2021/14904) («АмплиСенс», Россия).
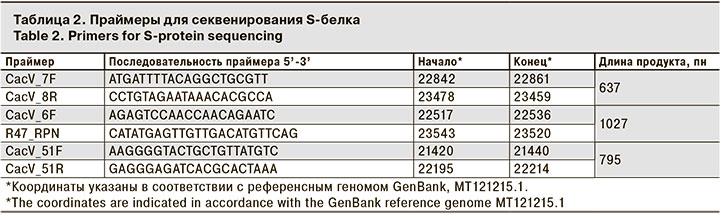
Для секвенирования по Сэнгеру проводили амплификацию фрагмента гена S-белка: брали 5 мкл кДНК, добавляли ее к смеси праймеров (табл. 2) (по 2,5 мкл праймера с концентрацией 10 пмоль/мкл) и дезоксинуклеотидов (2,5 мкл с концентрацией 4,4 мМ). Доводили объем смеси до 15 мкл деионизованной водой. К полученной смеси добавляли 10 мкл реагента «PCR-mixture-2 blue» («АмплиСенс», Россия), содержащего Taq-полимеразу в соответствующем буферном растворе. Реакцию амплификации проводили на амплификаторе T-100 (Bio-Rad, США). Программа амплификации: 95 °C – 45 с, затем 35 циклов 95 °C – 10 с, 52 °C – 30 с, 72 °C – 1 мин и финальная инкубация смеси при 72 °C в течение 3 мин. Затем проводили разделение продуктов реакции амплификации электрофорезом в 1% агарозном геле. Целевые фрагменты вырезали из геля и очищали с помощью набора реагентов DNA clean up MINI kit («Евроген», Россия).
Секвенирование ДНК проводили на автоматическом секвенаторе 3500x Genetic Analyzer (Applied Biosystems, CША), используя реагенты, рекомендованные фирмой-производителем.
Мутацию N501Y выявляли методом ПЦР-РВ с использованием собственной разработанной системы олигонуклеотидных праймеров и флуоресцентного зонда. Для проведения реакции брали 5 мкл кДНК, добавляли ее к смеси праймеров и зонда (по 2,5 мкл праймеров и зонда с концентрацией 10 пмоль/ мкл), дезоксинуклеотидов (2,5 мкл с концентрацией 4,4 мМ). Доводили объем смеси до 15 мкл деионизованной водой. К полученной смеси добавляли 10 мкл реагента, содержащего Taq-полимеразу в соответствующем буферном растворе. Реакцию проводили на амплификаторе ДТ-96 («ДНК-технология», Россия). Программа амплификации: 95 °C – 15 с, затем 45 циклов 95 °C – 10 с, 60 °C – 20 с. Полученные результаты анализировали в соответствии с инструкциями производителя в программе RealTime PCR v.7.9, поставляющейся с прибором («ДНК-технология», Россия).
Для обнаружения методом ИТ мутации N501Y в S-белке SARS-CoV-2, соответствующей замене A23063T в геноме коронавируса, был создан набор реагентов «АмплиСенс® SARS-CoV-2-N501Y-IT» (РУ № РЗН 2021/14904) с флуоресцентной детекцией продуктов амплификации в режиме реального времени. В состав набора входят 2 смеси праймеров, одна из которых подобрана к участку гена S длиной 200 нуклеотидов, содержащему замену A23063T. Внутренние праймеры FIP и BIP этой смеси несут на своих 5’-концах адениловый и тимидиловый нуклеотиды соответственно, что позволяет амплифицировать последовательность с SNP A23063T с помощью ИТ. В случае амплификации гена S вируса дикого типа формируются вторичные шпилечные структуры, несущие по одному неспаренному нуклеотиду на своих 3’-концах, что существенно затрудняет петлевую амплификацию, но не останавливает ее полностью (рисунок, см. на вклейке). Таким образом, для образцов вируса с мутацией N501Y характерны меньшие значения порогового цикла Ct на типирующей смеси праймеров по сравнению с образцами вируса дикого типа при одинаковой вирусной нагрузке.
Для оценки количества РНК SARS-CoV-2 в исследуемом образце используется вторая смесь праймеров, специфичных к консервативному локусу гена ORF1ab. Сопоставление значений пороговых циклов Ct для одной и той же пробы РНК вируса на 2 смесях позволяет сделать вывод о наличии (отсутствии) мутации N501Y. Для устранения влияния дополнительных мутаций под областями отжига праймеров и возможной разницы в скорости амплификации на первой и второй реакционных смесях в состав набора реагентов «АмплиСенс® SARS-CoV-2-N501Y-IT» входят ДНК-калибраторы с известной концентрацией. Интерпретация результатов анализа заключается в сопоставлении значений Ct исследуемого образца РНК и ДНК-калибраторов, полученных на обеих смесях праймеров внутри единой постановки ОТ-ИТ. Реакцию проводили на приборе ДТ-96 в соответствии с инструкциями производителя. Полученные данные анализировали в соответствии с инструкциями производителя в программе RealTime PCR v.7.9, поставляющейся с прибором («ДНК-технология», Россия). Обсчет полученных результатов проводили с применением электронного калькулятора на базе Microsoft® Excel.
Результаты
В рамках проведения работ по оценке генетического разнообразия SARS-CoV-2, циркулирующего на территории Москвы и Московской области, была разработана панель праймеров для секвенирования последовательности гена S-белка. Панель позволяет амплифицировать весь ген фрагментами от 500 до 1000 пн в зависимости от используемых комбинаций праймеров. Для рутинных исследований проводили секвенирование двух фрагментов S-гена с использованием пар праймеров CacV_51F/CacV_51R и CacV_7F/CacV_8R, соответствующих фрагментам белка с 55 по 255 а.к. и с 440 по 590 а.к. Это позволяет выявлять наличие мутаций delHV_69-70, delY_144, D80A, D138Y, G142D, del156-157, L452R, T478K, E484K, S494P, N501Y, A570D и др.
За период с 1 марта по 2 апреля 2021 г. в лабораторию поступило 1448 образцов назофарингеальных мазков от пациентов с подтвержденным диагнозом COVID-19. Для 372 образцов удалось провести генотипирование с помощью фрагментного секвенирования по методу Сэнгера. Генотипирование проводили по 2 локусам, в результате чего было выявлено наличие или отсутствие мутаций N501Y, E484K, D80A и делеций delHV_69-70 и delY144 и установлена принадлежность в геновариантам Альфа или Бета. Всего было выявлено 54 образца, относящихся к геноварианту Альфа, 7 – относящихся к геноварианту Бета и 25 – относящихся к геноварианту B.1.1.523. Последние содержат мутации S494P, E484K, F306L, делецию del156–158, но не содержат мутацию N501Y.
Для реализации скрининговых методов были разработаны методики выявления мутации N501Y в S-белке вируса SARS-Cov-2 методами ПЦР-РВ и ИТ.
Для ПЦР-РВ дополнительно была реализована возможность подтверждения результатов при помощи технологии пиросеквенирования. Такая комбинация позволяет использовать разработку как дополнение к существующей системе мониторинга геновариантов. В основе перечисленных методик – 45 циклов амплификации короткого фрагмента кДНК вируса (не более 120 пар оснований). Это имеет большое значение при амплификации образцов, содержащих небольшие количества вирусной РНК или кДНК, или в случаях, когда РНК или кДНК сильно фрагментированы.
Методом ПЦР-РВ было проанализировано 372 предварительно генотипированных образца для выявления мутации N501Y в гене S-белка коронавируса SARS-CoV-2. Из 61 образца, содержащего мутацию N501Y, было выявлено 59 мутаций, в том числе для 5 образцов геноварианта Бета, содержащих тандем мутаций E484K и N501Y. При тестировании 311 образцов кДНК в результате секвенирования методом ПЦР-РВ было установлено отсутствие мутации N501Y в 309 из них. Для всех образцов, относящихся к геноварианту B.1.1.523, содержащих мутации S494P и E484K, разработанная система корректно определила отсутствие мутации N501Y. Положительное прогностическое значение (ППЗ) для данного метода составляет 96,7%, отрицательное (ОПЗ) – 99,4%. Чувствительность и специфичность составляют 96,7 и 99,4% соответственно.
Методом петлевой ИТ было проанализировано 252 предварительно генотипированных образца. Из них 61 образец, в том числе 5 образцов геноварианта Бета, содержал мутацию N501Y. Из 61 образца с мутацией N501Y методом ИТ она была выявлена для 57, для 4 образцов был получен ложноотрицательный результат. Все остальные образцы (п = 191) методом ИТ все были определены как образцы без мутации. ППЗ для данного метода составило 93,2%, ОПЗ – 96,9%. Чувствительность и специфичность составили 91,2 и 97,9% соответственно.
Заключение
Проведено генотипирование по 2 локусам для 372 образцов мазков из рото- и носоглотки от пациентов с симптомами новой коронавирусной инфекции и подтвержденным методом ПЦР-РВ диагнозом COVID-19 с помощью фрагментного секвенирования по методу Сэнгера. Все они также были исследованы методом ПЦР-РВ на наличие мутации N501Y, а для 252 образцов ее наличие было определено методом ИТ. Продемонстрированы высокая чувствительность и специфичность метода ПЦР-РВ для выявления мутации N501Y коронавируса и более низкие соответствующие показатели для метода ИТ. На основании полученных данных можно рекомендовать скрининговые методы на основе ПЦР-РВ для выявления точечных мутаций коронавируса. Результаты исследования показывают перспективность разработки таких методов для анализа более широкого спектра мутаций, что с учетом возможностей мультиплексирования ПЦР позволит проводить генотипирование различных геновариантов с высокой точностью. Использование метода ИТ может быть хорошим решением при необходимости получения результата в более сжатые сроки.
Отдельно стоит отметить тот факт, что использование скрининговых методов генотипирования на основе ПЦР за счет получения коротких ампликонов длиной 100–200 пн. позволяет анализировать те образцы, секвенирование которых невозможно из-за небольшого количества генетического материала в пробе или из-за его низкого качества за счет сильного фрагментирования молекул РНК. Еще одним существенным преимуществом этих методов является значительное уменьшение временных затрат.
Следует добавить, что с помощью фрагментного или полногеномного секвенирования можно проанализировать в среднем от 50 до 80% проб с Ct меньше 20. Этот показатель сильно зависит от качества проб и соблюдения холодовой цепи при их хранении, транспортировке и обработке. Наши исследования показали, что образцы, которые не поддаются изучению методом секвенирования, можно исследовать методами на основе ПЦР в формате как ПЦР-РВ, так и ИТ. Дополнительно к 372 образцам с известным генотипом мы проанализировали методом ПЦР-РВ 424 несеквенированных образца. В результате было выявлено еще 54 образца, содержащих мутацию N501Y. Эти данные позволяют сделать вывод о том, что использование всех трех методов в арсенале лаборатории при проведении эпидемиологического мониторинга за распространением вируса позволяет получить более комплексную и исчерпывающую картину.


