В настоящее время к роду Ebolavirus, входящему в семейство Filoviridae, относят 5 видов вирусов: Заир (EBOV), Судан (SUDV), Рестон (RESTV), Бундибуджио (BDBV) и вирус леса Таи (TAFV) [1]. Из них 3 (EBOV, SUDV и BDBV) являются возбудителями острых лихорадок людей – лихорадок Эбола, летальность которых при отсутствии должного лечения варьирует, по разным оценкам, от 65–75% для EBOV до 55–58% для SUDV и BDBV [2–7]. Впервые человечество столкнулось с лихорадкой Эбола в 1976 г., когда сначала в Судане, а затем и в Республике Заир (ныне Демократическая Республика Конго) разразились вспышки, вызванные вирусами SUDV и EBOV. Наибольшее количество пострадавших было зарегистрировано в Республике Заир. Оно составило 318 человек [4]. В последующие годы спорадические вспышки лихорадки Эбола происходили на территории Центральной Африки, однако вспышка 1976 г. до недавнего времени считалась крупнейшей.
В феврале 2014 г. на территории Республики Гвинея возникла вспышка лихорадки Эбола, вызванная вирусом Заир (EBOV), которая распространилась на 5 стран, включая Гвинею, Либерию, Нигерию, Сенегал и Сьерра-Леоне [8]. Особенностью данной вспышки, переросшей в эпидемию, стало то обстоятельство, что территорией распространения инфекции стала Западная Африка, где ранее циркуляции вирусов рода Эбола не наблюдалось. По этой причине и в силу плохого оснащения лабораторной службы выявление возбудителя произошло с большой задержкой, что привело к запаздыванию соответствующих противоэпидемических мероприятий и распространению инфекции из сельской местности на городские территории с общим количеством населения около 25 млн человек. Поэтому, несмотря на то, что лихорадка Эбола считается природно-очаговым зоонозом, в настоящее время сложились условия, при которых данное заболевание распространяется в человеческой популяции без участия природного резервуара.
Несмотря на усилия мирового сообщества, число случаев заболевания лихорадкой Эбола в Западной Африке продолжает расти. По данным на 07.01.2015, общее число заболевших составило 20 747 человек, из них 8235 случаев закончились смертельным исходом [9]. Параллельно в Демократической Республике Конго возникла эндемическая вспышка лихорадки Эбола, также вызванная вирусом Заир (EBOV), который, однако, генетически отличен от изолятов, циркулирующих в Западной Африке [10].
 Тяжелая эпидемическая ситуация на Африканском континенте повышает вероятность завоза данного инфекционного агента на территорию Российской Федерации, что требует от компетентных служб выработки стратегии по недопущению распространения вируса Эбола Заир на территорию нашей страны. Одним из компонентов успешной реализации данной стратегии, безусловно, является наличие современных высокочувствительных и специфичных диагностических систем, позволяющих в сжатые сроки достоверно подтвердить наличие либо отсутствие вируса Эбола Заир в образцах от пациентов с подозрением на лихорадку Эбола. На сегодняшний день наиболее чувствительными, специфичными и требующими наименьших временных затрат при проведении анализа являются диагностические системы, принцип действия которых основан на методе ПЦР в реальном времени.
Тяжелая эпидемическая ситуация на Африканском континенте повышает вероятность завоза данного инфекционного агента на территорию Российской Федерации, что требует от компетентных служб выработки стратегии по недопущению распространения вируса Эбола Заир на территорию нашей страны. Одним из компонентов успешной реализации данной стратегии, безусловно, является наличие современных высокочувствительных и специфичных диагностических систем, позволяющих в сжатые сроки достоверно подтвердить наличие либо отсутствие вируса Эбола Заир в образцах от пациентов с подозрением на лихорадку Эбола. На сегодняшний день наиболее чувствительными, специфичными и требующими наименьших временных затрат при проведении анализа являются диагностические системы, принцип действия которых основан на методе ПЦР в реальном времени.
Цель работы – разработка диагностической системы в формате ОТ-ПЦР с гибридизационно-флуоресцентной детекцией сигнала для выявления РНК вируса Эбола Заир в биологических образцах и определение ее аналитических характеристик.
Материалы, методы и результаты
Дизайн олигонуклеотидных праймеров и зонда
В качестве мишени для олигонуклеотидных праймеров и зонда выбрали консервативный фрагмент первичной нуклеотидной последовательности гена полимеразы (L-ген) длиной 372 н. т. (позиции 13997–14368 н. т. референсного сиквенса EBOV, номер доступа в GenBank NCBI – HQ613402), выявленный путем выравнивания всех представленных в GenBank NCBI первичных нуклеотидных последовательностей вируса Эбола Заир (рис. 1). Выравнивание провели с помощью пакета программ Vector NTI Advanced v.11 («Invitrogen», USA). Выбор столь длинного фрагмента в качестве мишени обусловлен желанием иметь возможность подтверждения положительных результатов секвенированием полученного ПЦР-продукта ввиду отсутствия в момент разработки информации о первичных нуклеотидных последовательностях изолятов вируса Эбола Заир, полученных в ходе эпидемии 2014 г. Ретроспективный анализ показал 100% идентичность структур праймеров и зонда первичным нуклеотидным последовательностям изолятов из Западной Африки (текущая эпидемия) [11], а также изолятов из ДРК, полученных в 2014 г. [10].
Дизайн праймеров и зонда проводили с учетом общих требований, предъявляемых к олигонуклеотидным праймерам и зондам типа TaqMan при проектировании тест-систем в формате ПЦР в реальном времени [12–14]. Структуры праймеров и зонда представлены в табл. 1. Оценку температуры плавления праймеров рассчитывали при помощи программы Oligonucleotide Properties Calculator [15]. Термодинамические характеристики зонда и возможность образования вторичных структур оценивали с помощью программ Oligonucleotide Properties Calculator и MFOLD [16]. Для снижения уровня фонового сигнала на 3’-конец зонда добавили искусственную последовательность из трех нуклеотидных остатков, что позволило сместить термодинамическое равновесие в сторону образования «шпилечной» вторичной структуры (рис. 2). При синтезе зонда в качестве флуорофора использовали R6G (родамин 6 Ж), а в качестве тушителя – BHQ-1 (black hole quencher1).
 Создание положительного контрольного образца
Создание положительного контрольного образца
Ввиду отсутствия генетического материала вируса Эбола Заир создание положительных контрольных образцов осуществляли синтетическим методом на основе ампликона, включающего в себя таргетную область и фланкирующие последовательности нуклеотидов. Ампликон получали методом «степ-аут» ПЦР. Для этого синтезировали 8 пар олигонуклеотидов прямой и обратной ориентации со средней длиной 65 н. т. и перекрытием 25 н. т. Дизайн олигонуклеотидов осуществляли на основании референсного сиквенса EBOV (код доступа в GenBank NCBI – HQ613402).
Для получения промежуточных продуктов амплификации готовили 25 мкл реакционной смеси, содержащей 0,5 мкл соответствующего ПЦР-продукта, по 50 пмоль прямого и обратного олигонуклеотидов, 2,5 мкл дНТФ (1,76 мM, «АмплиСенс», Россия), 10 мкл ПЦР-смеси-2 blue (ЦНИИЭ, Россия). Термоциклирование проводили в следующем режиме: 94 °C – 45 с, 94 °C – 20 с, 50 °C – 30 с (n = 5); 72 °C – 10 мин. Реакцию проводили с использованием термоциклера Maxygen II («Axygen», США). Результат амплификации контролировали электрофоретическим методом в 1,5% агарозном геле с добавлением бромистого этидия. Конечный ПЦР-продукт очищали с помощью набора MiniElute Gel Extraction Kit («QIAGEN», Германия), лигировали в плазмидный вектор pGEM-T («Promega», США) под контролем промотора T7 РНК полимеразы и трансформировали им Escherichia coli (штамм XL1-Вlue) [17]. Рекомбинантные плазмиды из индивидуальных клонов очищали с помощью набора Plasmid Miniprep Kit («Axygen», США) и проверяли их на наличие правильной ориентации и отсутствие мутаций в области посадки праймеров и зонда. Проверку осуществляли методом Сэнгера с помощью прибора для автоматического капиллярного секвенирования ABI PRISM 3500xl («Applied Biosystems», США). Плазмиды с прямыми вставками использовали для получения рекомбинантной РНК на основе MS2 фага в защитной белковой оболочке [18]. Полученный продукт обрабатывали ДНКазой I («Fermentas», Латвия) для удаления остатков ДНК, измеряли концентрацию, разбавляли в стабилизирующем растворе RNA-later («Life Technologies», США) и использовали далее в качестве положительного защищенного РНК-контроля (ПКО). Отсутствие в препарате ПКО остаточной ДНК после обработки контролировали с помощью специфических праймеров и зонда в ходе проведения ПЦР в реальном времени (без стадии обратной транскрипции). Измерение концентрации рекомбинантного контроля осуществляли на приборе QX100 system («Bio-Rad», США) с помощью набора One-Step ddPCR Supermix for Probes Kit («Bio-Rad», США) и специфических праймеров и зонда (см. тaбл. 1).
Состав реакционной смеси и режим амплификации
Реакцию ОТ-ПЦР в реальном времени проводили в объеме 25 мкл. Реакционная смесь содержала следующие компоненты: 10 мкл РНК-пробы, 1 мкл праймера rt_EBOV-f (5 пмоль/мкл), 1 мкл праймера rt_EBOV-r (5 пмоль/мкл), 1 мкл зонда EBOV_prb (5 пмоль/мкл), 2,5 мкл дНТФ (1,76 мМ, «АмплиСенс», Россия), 5 мкл смеси RT-PCR mix2 FEP/FRT («АмплиСенс», Россия), 0,25 мкл MMLV-ревертазы («АмплиСенс», Россия), 0,25 мкл RTG-mix2 (ЦНИИЭ, Россия) и 0,5 мкл TaqF-полимеразы (ЦНИИЭ, Россия). Реакцию осуществляли в приборе для ПЦР в реальном времени Rotor-Gene 6000 («QIAGEN», Германия) в следующем режиме: 50 °C – 15 мин, 95 °C – 15 мин; 95 °C – 10 с, 55 °C – 20 с, 72 °C – 15 с (n = 45). Детекцию проводили при 55 °C на канале Yellow.
Аналитическая чувствительность и специфичность
Специфичность диагностической системы оценивали с помощью кДНК из высокотитражных культур 23 видов вирусов, принадлежащих к 10 различным семействам (табл. 2), а также 30 образцов клинического материала (плазма) от клинически здоровых людей. В результате исследования ложноположительных результатов не зафиксировано.
Для оценки аналитической чувствительности готовили с десятикратным шагом разведения (ПКО) на плазме, выделяли с помощью набора для экстракции нуклеиновых кислот Рибо-преп («АмплиСенс», Россия) согласно рекомендациям производителя и исследовали с помощью диагностической системы для выявления минимального порогового разведения, при котором образец детектируется как положительный. По данным исследования, аналитическая чувствительность системы составила 2 × 103 ГЭ/мл. Аналитическую чувствительность оценивали также с помощью стандартного диагностического образца вируса Эбола Заир, предоставленного институтом Роберта Коха (Берлин, Германия). Препарат представляет собой инактивированные и лиофилизированные вирусные частицы известной концентрации и рекомендован ВОЗ как тест-объект для оценки работоспособности систем, используемых для диагностики вируса Эбола Заир. Согласно прилагаемому паспорту, разведенный в 100 мкл стандартный диагностический образец содержал инактивированные вирусные частицы в концентрации 2 × 107 ГЭ/мл. Из полученного раствора методом последовательных разведений готовили образцы стандартного препарата в концентрациях 5 × 103, 103 и 102 ГЭ/мл. Для приготовления разведений использовали буфер ТЕ (ЦНИИЭ, Россия). Из полученных разведений выделяли вирусную РНК с помощью набора для выделения Viral RNA Kit («QIAGEN», Германия) согласно рекомендациям производителя и исследовали с помощью разработанной системы для выявления минимального разведения, при котором стандартный образец детектируется как положительный. По данным исследования, аналитическая чувствительность системы, оцененная данным способом, составила 103 ГЭ/ мл.
Нормализованные кривые флуоресцентного сигнала, полученные при оценке аналитической чувствительности диагностической системы, представлены на рис. 3 (А, Б).
Обсуждение
Была создана первая отечественная диагностическая система в формате ОТ-ПЦР в реальном времени для диагностики вируса Эбола Заир. Система содержит полный комплект реагентов для проведения анализа, включая реагенты для выделения РНК. Наличие защищенного ПКО позволяет контролировать качество всех этапов проведения анализа, в том числе и этап выделения нуклеиновых кислот.
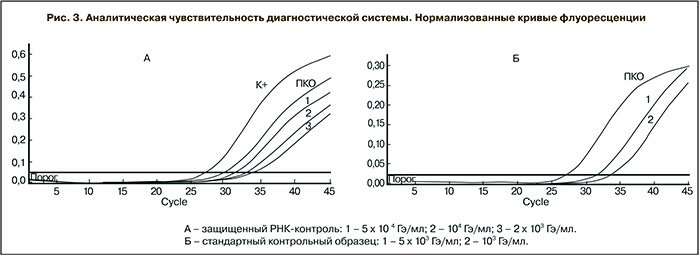
Аналитическая чувствительность системы, оцененная двумя способами, совпадает в пределах погрешности измерения методом ПЦР в реальном времени и составляет 1–2 × 103 ГЭ/мл. Полученное пороговое значение чувствительности аналогично таковым для зарубежных аналогов и является стандартным для диагностических систем в формате ОТ-ПЦР в реальном времени.
К сожалению, большинство ранее разработанных диагностических систем оказалось малопригодно для выявления изолятов из Западной Африки, поскольку те имеют замены в областях, выбранных разработчиками в качестве мишеней для амплификации [11]. Данное обстоятельство создало определенные трудности в организации адекватной лабораторной диагностики лихорадки Эбола, чем, возможно, и объясняется невысокий процент лабораторно подтвержденных случаев заболевания.
За счет удачного выбора таргетной области для амплификации разработанная система способна одинаково успешно выявлять изоляты, циркулирующие как в Западной, так и в Центральной Африке. Разработанная диагностическая система поможет не только сохранить санитарно-эпидемическое благополучие в отношении вируса Эбола Заир на территории Российской Федерации, но и будет способствовать ликвидации продолжающейся эпидемии лихорадки Эбола на Африканском континенте.
Выводы
Разработана диагностическая система в формате ОТ-ПЦР в реальном времени для выявления РНК вируса Эбола Заир.
Результаты испытаний показали, что разработанная система по своим аналитическим характеристикам не уступает ведущим зарубежным аналогам и является надежным инструментом диагностики лихорадки Эбола, вызванной вирусом Заир.


